Command Palette
Search for a command to run...
在线教程|算力成本骤降!苹果基于流匹配方法,推出蛋白质折叠模型 Ml-simplefold

2025 年 9 月,苹果公司推出了轻量级蛋白质折叠预测 AI 模型 Ml-simplefold 。作为第一个基于流匹配的蛋白质折叠模型,在 CAMEO22 和 CASP14 等权威基准测试中,SimpleFold 在降低计算成本后,仍显示出与 AlphaFold2 、 RoseTTAFold2 等顶尖模型相当的性能。同时其小规模版本 SimpleFold-100M 的表现同样具有竞争力。
传统的蛋白质折叠模型虽然精度可观,但依赖大量领域特定的架构设计与手工特征。模型中常用的三角更新模块、显式对表示机制及多重训练目标使训练算力成本高昂,同时架构、硬件缺乏可扩展性,难以支持多样化结构的生成或集成预测。
针对传统方法的通用型缺陷,SimpleFold 提出了一个纯基于 Transformer 的通用生成式框架,打破了蛋白质折叠模型对复杂架构的依赖:
* 基于流匹配(Flow Matching)技术,跳过多序列比对(MSA)等复杂模块,直接从随机噪声生成蛋白质的三维结构,大幅降低计算成本;
* 采取摒弃三角更新和对表示等特定模块的通用架构,仅使用标准 Transformer,并通过自适应层增强结构感知能力;
* 加入结构约束,使模型在生成蛋白质三维结构时具备更高的灵活性与物理一致性,在 9M 结构数据上训练的 3B 参数模型可在消费级硬件上流畅运行,大幅降低计算门槛。
「Ml-simplefold:轻量级蛋白质折叠预测 AI 模型」现已上线 HyperAI 超神经官网(hyper.ai)的「教程」板块,一键运行即可体验最新的蛋白质生成工具~
教程链接:
Demo 运行
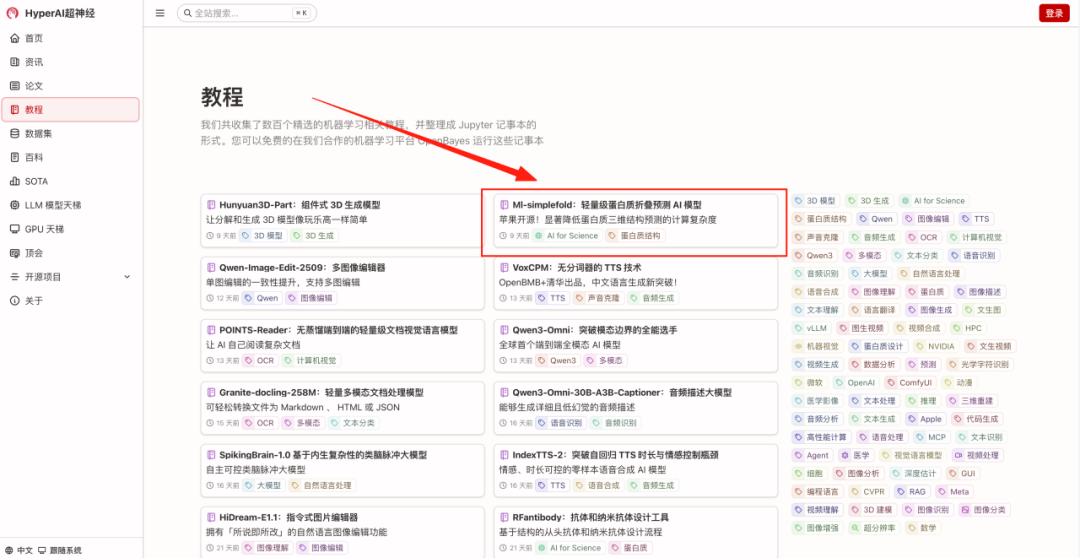
- 在浏览器输入网址 hyper.ai,进入首页后,点击「教程」页面,并选择「基于流匹配的蛋白质折叠模型」,点击「在线运行此教程」。
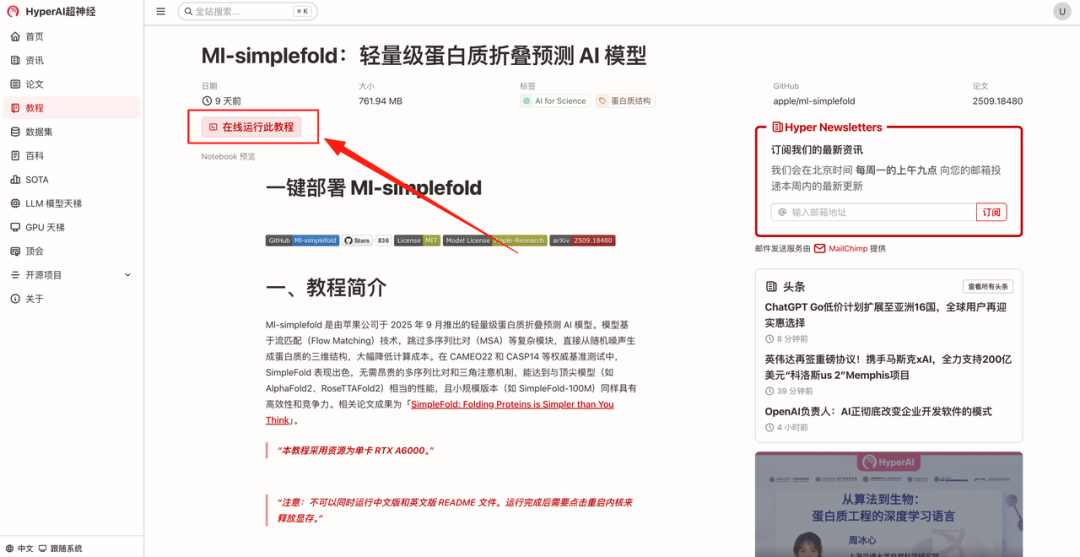
- 页面跳转后,点击右上角「克隆」,将该教程克隆至自己的容器中。
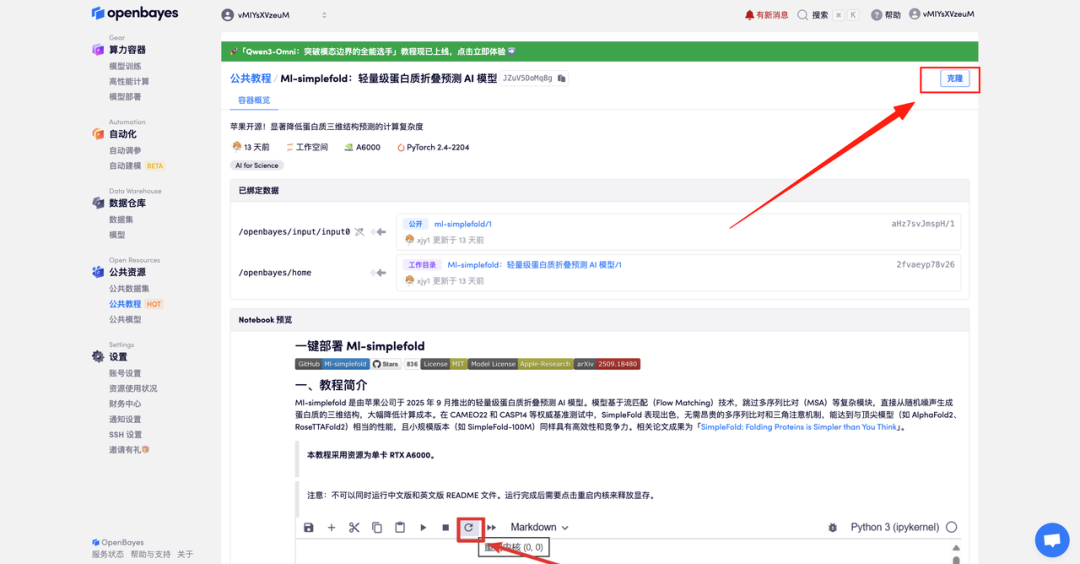
- 选择「NVIDIA RTX A6000 48GB」以及「PyTorch」镜像,并点击「继续执行」。 OpenBayes 平台提供了 4 种计费方式,大家可以按照需求选择「按量付费」或「包日/周/月」。新用户使用下方邀请链接注册,可获得 4 小时 RTX 4090 + 5 小时 CPU 的免费时长!
HyperAI 超神经专属邀请链接(直接复制到浏览器打开):
https://openbayes.com/console/signup?r=Ada0322_NR0n
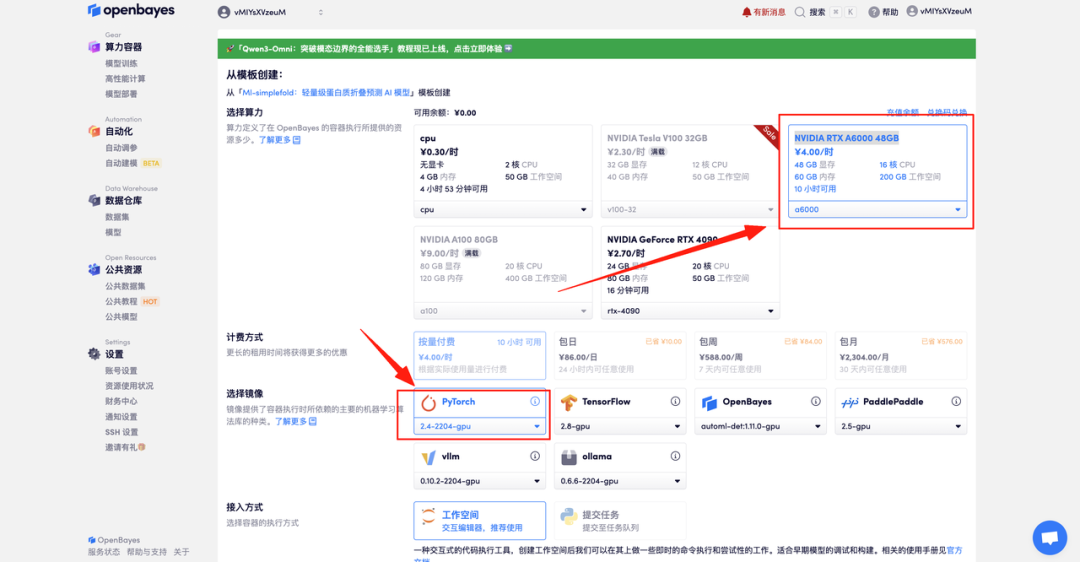
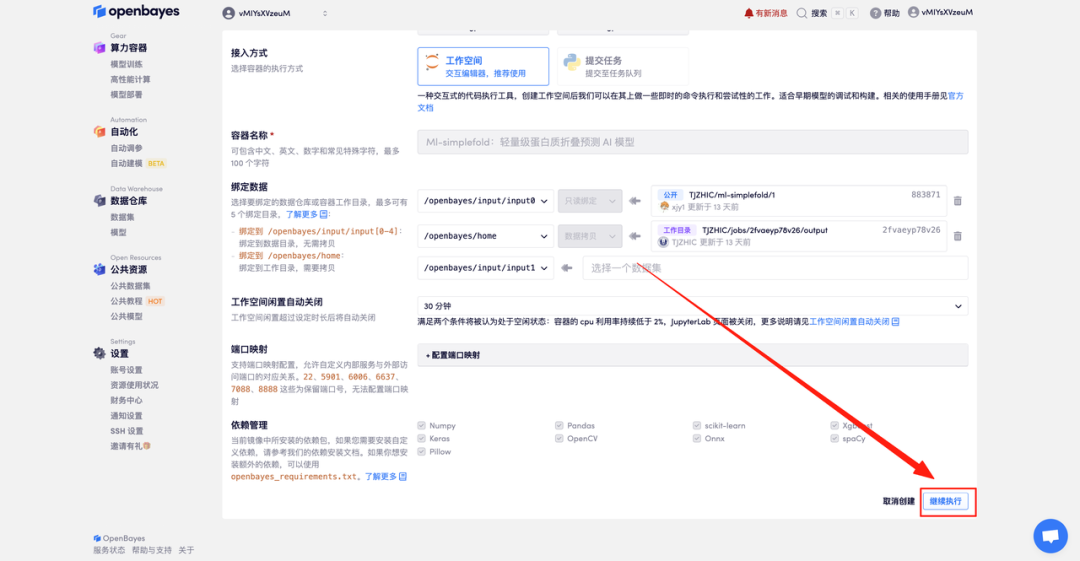
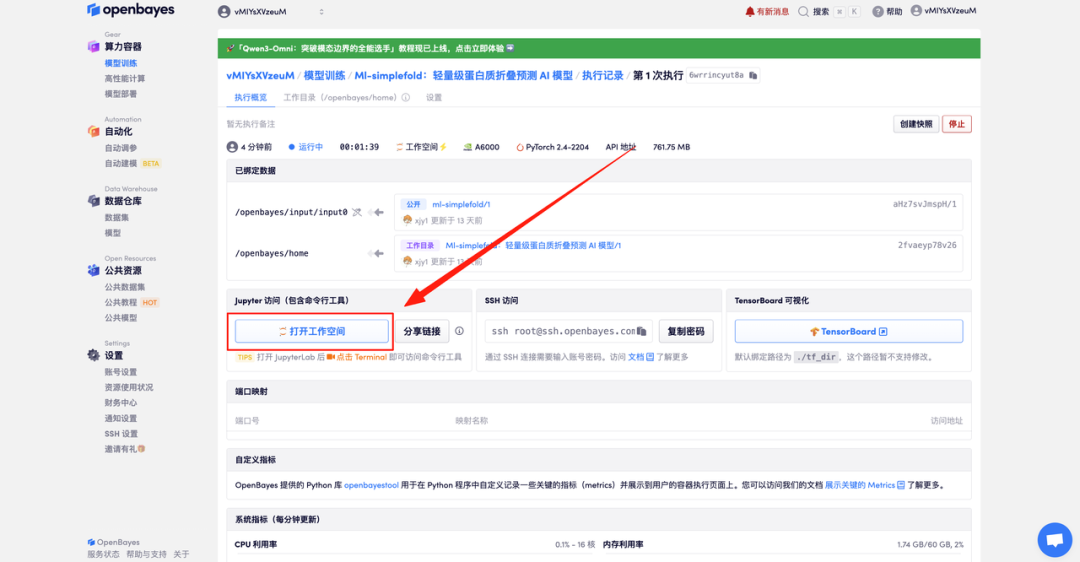
4. 等待分配资源,首次克隆需等待约 2 分钟左右的时间。当状态变为「运行中」后,点击「打开工作空间」即可跳转至 Demo 页面。
效果演示
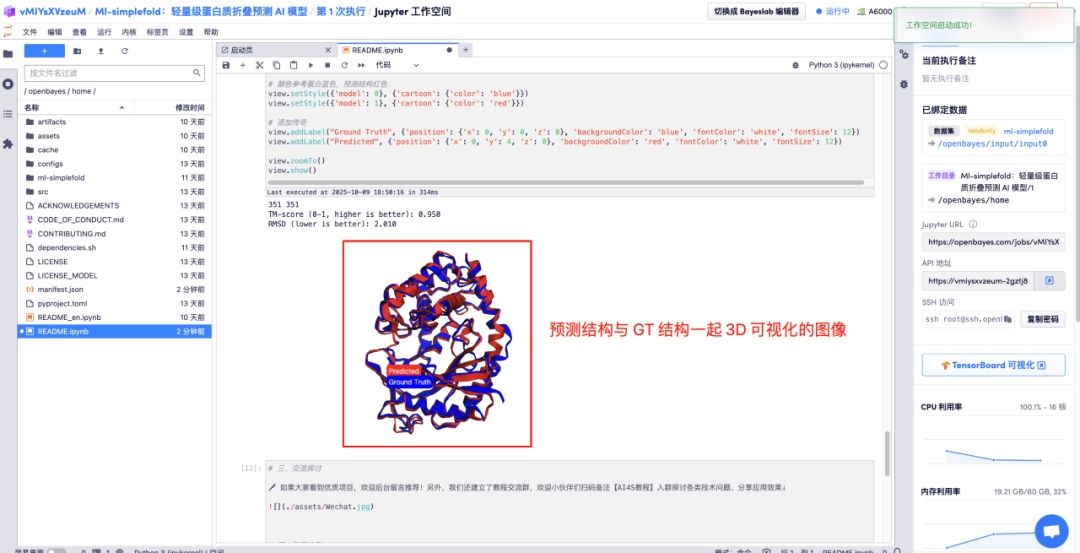
以下为 Ml-simplefold 使用页面,点击「README」即可跳转至生成界面。
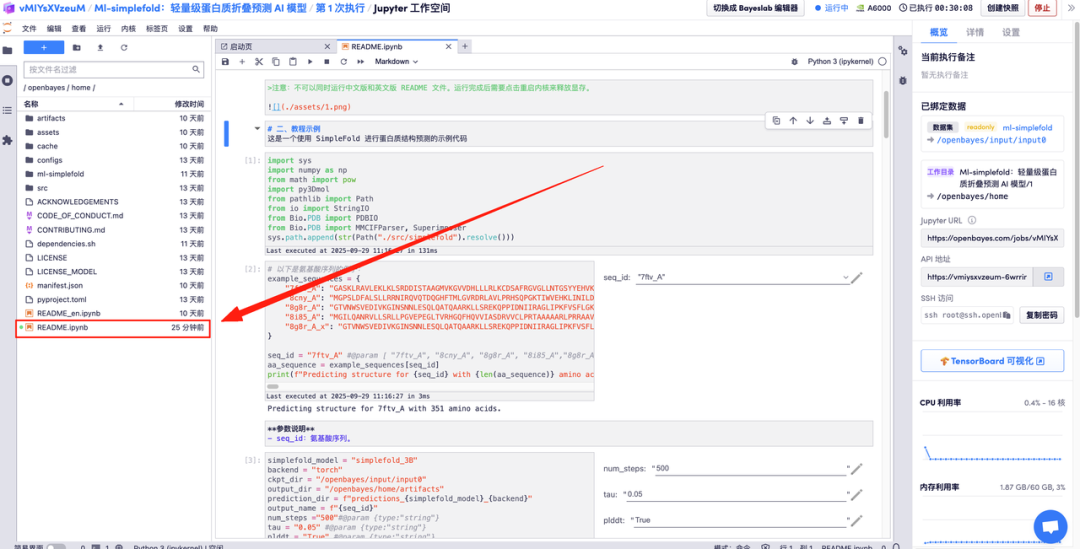
在对应区域分别输入氨基酸参数说明,点击运行,即可得到预测的蛋白质文件、全原子结构和与 GT 结构合并的 3D 可视化图像。
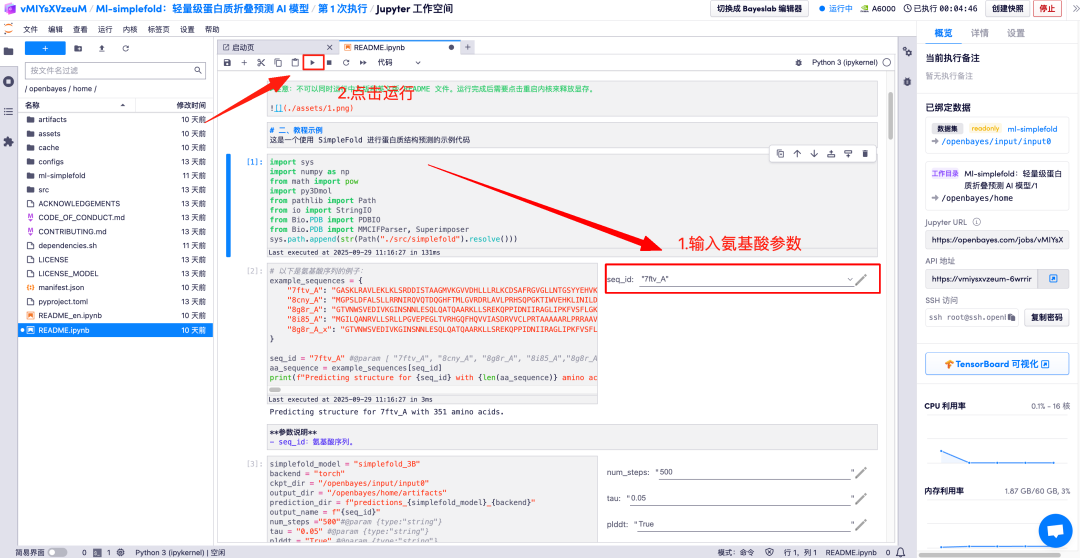
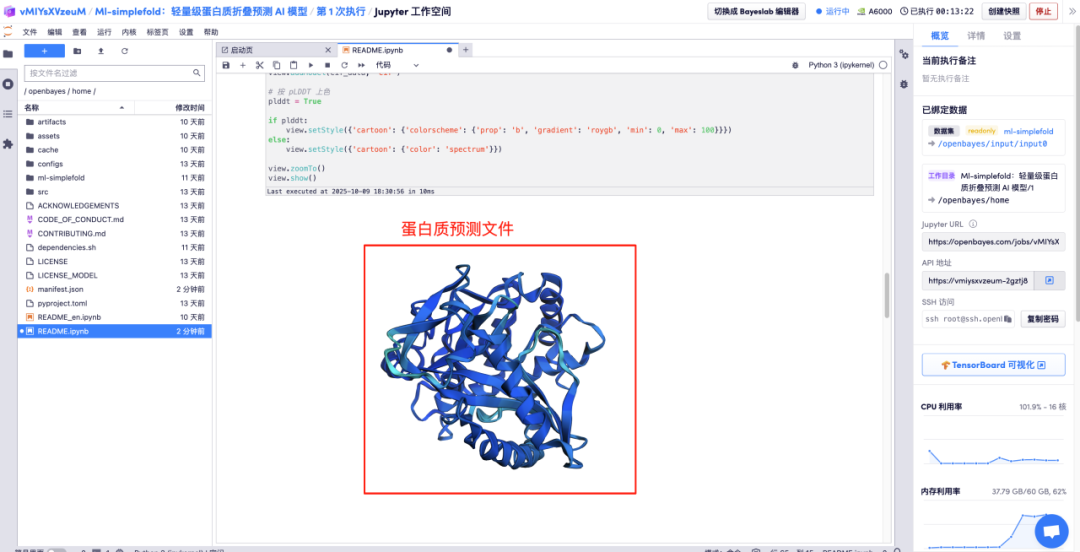
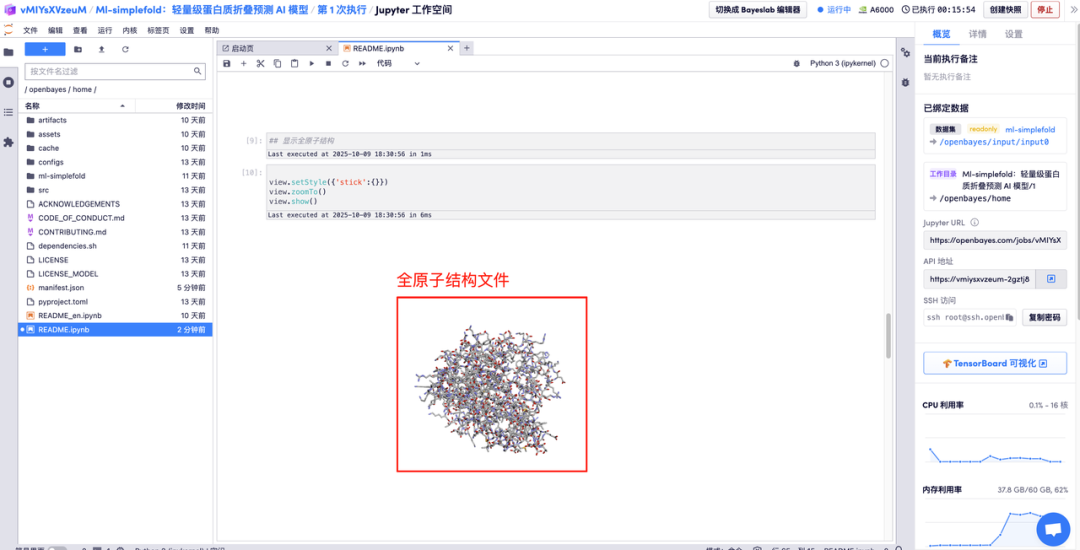
以上就是 HyperAI 超神经本期推荐的教程,欢迎大家前来体验!
教程链接: